Problem 10: Neo-Darwinismen har en lang historie med unøyaktige prediksjoner om vestigale organer og 'søppel-DNA'
Oversatt herfra.
 I flere tiår har evolusjonister hevdet at kroppene og genomene våre er fulle av ubrukelige deler og genetisk materiale - 'vestigiale' organer - som viser at livet er et resultat av eoner av ikke-styrt evolusjon. Under Scopes-rettssaken i 1925 hevdet evolusjonærbiologen Horatio Hackett Newman at det er over 180 vestigiale organer og strukturer i menneskekroppen, 'tilstrekkelig til å gjøre en mann til et veritabelt vandrende museum av antikviteter.' (157)
I flere tiår har evolusjonister hevdet at kroppene og genomene våre er fulle av ubrukelige deler og genetisk materiale - 'vestigiale' organer - som viser at livet er et resultat av eoner av ikke-styrt evolusjon. Under Scopes-rettssaken i 1925 hevdet evolusjonærbiologen Horatio Hackett Newman at det er over 180 vestigiale organer og strukturer i menneskekroppen, 'tilstrekkelig til å gjøre en mann til et veritabelt vandrende museum av antikviteter.' (157)
Over tid har imidlertid ikke disse spådommene av vestigiale kroppsdeler og ubrukelig DNA vist seg sanne. I det forskere har lært mer og mer om biologiens produkter, har viktige funksjoner og formål blitt oppdaget for disse såkalte vestigiale strukturer. Faktisk rapporterte journalen New Scientist i 2008, siden professor Newman-dagene, at listen over vestigiale organer 'vokste og deretter krympet igjen' til det punktet at 'biologer i dag er svært forsiktige med å snakke om vestigiale organer i det hele tatt.' (158) Strukturer som tidligere var - og feilaktig - ble betraktet å være vestigiale, inkluderer:
Mandlene: En gang ble de rutinemessig fjernet. Nå er det kjent at de tjener en hensikt i lymfesystemet for å bekjempe infeksjoner. (159)
 Halebenet (coccyx ): Mange evolusjonister hevder fortsatt at dette 'henger igjen' fra haler til våre antatte primater forfedre, (160), men det er faktisk en viktig del av skjelettet vårt, som brukes til å feste muskler, sener og leddbånd som støtter beinene i bekkenet vårt.
Halebenet (coccyx ): Mange evolusjonister hevder fortsatt at dette 'henger igjen' fra haler til våre antatte primater forfedre, (160), men det er faktisk en viktig del av skjelettet vårt, som brukes til å feste muskler, sener og leddbånd som støtter beinene i bekkenet vårt.
Skjoldbruskkjertelen: Denne kjertelen i nakken ble en gang antatt ikke å ha noen hensikt, og ble ignorert eller endog ødelagt av medisinske leger som opererer under falske darwinistiske antagelser. Nå vet forskere at det er viktig å regulere metabolismen.
Blindtarmen: Darwinistiske forskere har hevdet at blindtarmen er et 'vestigalt fra våre plantelevende forfedre' (161) og over evolusjonens eoner har dens funksjon hos mennesker blitt redusert eller gått tapt. Men det er nå kjent at vedhenget utfører viktige funksjoner, for eksempel å gi et lagringsplass til gunstige bakterier, produsere hvite blodlegemer, og spille viktige roller under fosterutvikling. (162) I lys av dette beviset bemerket Duke University immunolog William Parker at "mange biologi tekster i dag fortsatt refererer til blindtarmen som et 'vestigial organ', men 'det er på tide å rette opp lærebøkene'. (163)
Til tross for den dårlige historikken i å påstå at organer var vestigiale, har evolusjonære biologer anvendt samme type tenkning på våre genomer. Mange har postulert at den tilfeldige naturen til mutasjoner ville fylle våre genom med ubrukelig genetisk søppel, kalt 'søppel DNA'. Denne hypotesen ble tilsynelatende bekreftet da det ble oppdaget at bare 2% av det menneskelige genomet kodet for proteiner, og etterlot de andre 98% uforklarte.
 Mange forskere som tjener som talsmenn for evolusjonær biologi har hevdet at dette beviset 'lukker saken', som bevis for darwinistisk evolusjon:
Mange forskere som tjener som talsmenn for evolusjonær biologi har hevdet at dette beviset 'lukker saken', som bevis for darwinistisk evolusjon:
*Brown University Evolutionary Biologist Kenneth Miller argumenterer for at "det menneskelige genomet er full av pseudogener, genfragmenter, 'foreldreløse' gener, 'junk' DNA og så mange gjentatte kopier av meningsløse DNA-sekvenser at det ikke kan tilskrives noe som ligner intelligent design ." (164)
*Richard Dawkins skriver også at "kreasjonister kan bruke litt tid på alvorlig å spekulere om hvorfor Skaperen burde bry seg med å forsøple gener med ikke-oversatte pseudogener og søppel-tandem gjentatt DNA." (165)
*I sin bok fra 2006 'The Language of God', hevdet Francis Collins at ca '45 prosent av det menneskelige genom' består av 'genetisk flotsam og jetsam'. (166) (Flotsam og jetsam er selvsagt ubrukelig søppel som flyter i havet.) I det han ligner mye på Dawkins, gjør han implikasjonene klare: "Med mindre man er villig til å ta stillingen at Gud har plassert [delt funksjonsløst repeterende DNA] i disse nøyaktige posisjoner for å forvirre og villede oss, er konklusjonen om en felles stamfar for mennesker og mus nesten uunngåelig." (167)
Problemet med disse argumentene er ikke så mye teologiske som de er vitenskapelig: Mange eksempler på funksjonalitet har blitt oppdaget for såkalt junk DNA.
Biolog Richard Sternberg undersøkte litteraturen og fant omfattende bevis på funksjonalitet for repeterende DNA. Han skrev i 'Annals of the New York Academy of Sciences' , fant at funksjoner for gjentakelser inkludert dannelse av høyere ordens nukleare strukturer, sentromerer, telomerer og kjerne-sentre for DNA-metylering. Repeterende DNA ble funnet å være involvert i celleproliferasjon, cellulære stressresponser; gen-oversettelse og DNA-reparasjon. (168) Sternberg konkluderte med at "de selviske [søppel] DNA fortellingene og lignende rammeverk må slå lag med de andre 'ikonene' for den nydarwinistiske evolusjonsteorien, som til tross for sin variasjon med empiriske bevis fortsatt overlever i litteraturen." (169)
Andre undersøkelser har fortsatt å avdekke funksjoner for ulike typer repeterende DNA, inkludert SINE (170), LINE (171) og Alu elementer (172). En artikkel foreslo endog at repeterende Alu- sekvenser kan være involvert i "utviklingen av høyere hjernefunksjoner" hos mennesker (173). Mange andre funksjoner har blitt oppdaget for ulike typer ikke-proteinkodende-DNA, inkludert:
reparere DNA (174); assistere i DNA-replikasjon (175); regulere DNA transkripsjon (176); hjelp i folding og vedlikehold av kromosomer (177); kontrollere RNA-redigering og spleising (178); bidrag til å bekjempe sykdom (179); regulering av embryologisk utvikling (180).
 Sternberg, sammen med University of Chicago genetikeren James Shapiro, forutsa i 2005 i tidsskriftet 'Cytogenetic and Genome Research' at 'en dag vil vi tenke på det som pleide å bli kalt 'junk DNA' som en kritisk del av virkelig 'ekspert' cellulære kontroll-regimer'. (181)
Sternberg, sammen med University of Chicago genetikeren James Shapiro, forutsa i 2005 i tidsskriftet 'Cytogenetic and Genome Research' at 'en dag vil vi tenke på det som pleide å bli kalt 'junk DNA' som en kritisk del av virkelig 'ekspert' cellulære kontroll-regimer'. (181)
Dagen forutsatt av Sternberg og Shapiro kan ha kommet raskere enn de forventet. I september 2012 rapporterte journalen Nature resultatene fra et årelangt forskningsprosjekt, med over 400 internasjonale forskere som studerte funksjonene til ikke-kodende DNA hos mennesker. Kalt ENCODE-prosjektet, rapporterte dets samling av 30 banebrytende artikler at 'den store majoriteten' av genomet har funksjon. Lederartikkelen som rapporterte ENCODEs resultater erklærte:
-Disse dataene gjorde det mulig for oss å tildele biokjemiske funksjoner til 80% av genomet, spesielt utenfor de godt studerte proteinkodende områdene. (182)
 Ewan Birney, ENCODEs ledende analysekoordinator kommenterte i Discover Magazine at siden ENCODE så på bare 147 typer celler, og menneskekroppen har noen få tusen, "Det er sannsynlig at 80 prosent vil øke til 100 prosent." (183) I samme artikkel sitert Tom Gingeras, en seniorforsker med ENCODE, og bemerker at "nesten hvert nukleotide er knyttet til en eller annen funksjon, og vi vet nå hvor de er, hva som binder til dem, hva deres relasjoner er, og mer." En annen Nature kommentar bemerket at "80% av genomet inneholder elementer knyttet til biokjemiske funksjoner, og senker den allment holdte oppfatningen om at det menneskelige genomet hovedsakelig er 'junk DNA'. (185) Discover Magazine sa det slik: "Hovedpoenget er: Det er ikke" søppel ". (186)
Ewan Birney, ENCODEs ledende analysekoordinator kommenterte i Discover Magazine at siden ENCODE så på bare 147 typer celler, og menneskekroppen har noen få tusen, "Det er sannsynlig at 80 prosent vil øke til 100 prosent." (183) I samme artikkel sitert Tom Gingeras, en seniorforsker med ENCODE, og bemerker at "nesten hvert nukleotide er knyttet til en eller annen funksjon, og vi vet nå hvor de er, hva som binder til dem, hva deres relasjoner er, og mer." En annen Nature kommentar bemerket at "80% av genomet inneholder elementer knyttet til biokjemiske funksjoner, og senker den allment holdte oppfatningen om at det menneskelige genomet hovedsakelig er 'junk DNA'. (185) Discover Magazine sa det slik: "Hovedpoenget er: Det er ikke" søppel ". (186)
Mens det fortsatt er mye vi ikke vet om genomet, peker forskningens trendlinje klart i en retning: Jo mer vi studerer genomet, desto mer oppdager vi funksjon for ikke-kodende DNA. Men det nå tvilsomme 'junk-DNA' -paradigmet ble født og avlet inne i det evolusjonære paradigmet, basert på ideen om at vårt genom ble bygget gjennom tilfeldige mutasjoner. Ja, noen få 'tvilsomme' biologer våget å søke funksjon for ikke-kodende DNA, men det darwinistiske 'junk DNA' -synet på genetikk har generelt hindret vitenskapens fremgang, slik det ble innrømmet av en 2003-artikkel i Science :
-Selv om det er fengende, har termen 'junk DNA' i mange år hindret vanlige forskere fra å studere ikke-kodende DNA. Hvem, bortsett fra et lite antall genomiske 'landstrykere', vil like grave gjennom genomisk søppel?
Imidlertid er det i vitenskap som i dalgliglivet noen landstrykere som, i fare for å bli latterliggjort, utforsker upopulære territorier. På grunn av dem begynte synet på 'søppel DNA', spesielt repeterende elementer, å endres tidlig på 1990-tallet. Nå, betrakter flere og flere biologer repeterende elementer som en genomisk skatt. (187)
Til tross for utbredte darwinske antagelser om det motsatte, konkluderte artikkelen at 'repeterende elementer ikke er ubrukelig søppel-DNA, men snarere viktige, integrerte komponenter' (188) av dyre genomer. Studier tyder på at disse lange strekkene av ikke-kodende DNA mellom gener 'utgjør et viktig lag av genom-regulering over et bredt spekter av arter.' (189)
Lik repetitive elementer, er en annen type 'søppel' DNA for hvilken funksjon oppdages, pseudogener. Pseudogener antas å være kopier av engang funksjonelle gener som har blitt inaktivert gjennom mutasjoner. En artikkel i Science Signaling observerer at 'pseudogener lenge har blitt avvist som søppel-DNA' (190) men bemerker:
-Nylige fremskritt har fastslått at DNA fra et pseudogen, RNA transkribert fra et pseudogen eller proteinet som er oversatt fra et pseudogen, kan ha flere forskjellige funksjoner, og at disse funksjonene kan påvirke ikke bare deres opphavelige gener, men også ikke-relaterte gener. Derfor har pseudogener dukket opp som en tidligere ikke-verdsatt klasse av sofistikerte modulatorer av genuttrykk, med et mangesidig engasjement i patogenesen for menneskelig kreft. (191)
 Faktisk har funksjoner for mange pseudogener blitt oppdaget; (192) ENCODE-prosjektet alene fant over 850 pseudogener som er 'transkribert og forbundet med aktivt kromatin.' (193) Men hva gjør disse pseudogenene? En 2011-artikkel i journalen RNA argumenterer igjen for at de kan regulere uttrykket av gener:
Faktisk har funksjoner for mange pseudogener blitt oppdaget; (192) ENCODE-prosjektet alene fant over 850 pseudogener som er 'transkribert og forbundet med aktivt kromatin.' (193) Men hva gjør disse pseudogenene? En 2011-artikkel i journalen RNA argumenterer igjen for at de kan regulere uttrykket av gener:
-Pseudogener har lenge vært merket som 'junk' DNA, mislykkede kopier av gener som oppstår under utviklingen av genomene. Men de siste resultatene utfordrer dette klengenavnet; Faktisk synes noen pseudogener å ha potensiale til å regulere sine proteinkodende fettere. (194)
På samme måte erklærte en artikkel i 2012 i tidsskriftet 'RNA Biology' at "Pseudogener lenge ble betraktet som søppel genomisk DNA", men "pseudogen regulering er utbredt" (195) i komplekse multicellulære organismer. Artikkelen foreslo at 'høy overflod og bevaring av pseudogener i en rekke arter, indikerer at selektivt trykk bevarer disse genetiske elementene, og foreslår at de faktisk kan utføre viktige biologiske funksjoner." (196)
Pseudogener tjener som et annet godt eksempel på hvordan darwinske biologer har antatt at en type ikke-kodende DNA de ikke forsto, var funksjonsløst genetisk søppel, og dermed ignorerte de deres funksjoner. Faktisk forklarer ovennevnte papir i RNA Biology at en grunn til at evolusjonister har vært så trege i å forlate antakelsen om at pseudogener er søppel, er fordi deres funksjoner er vanskelige å oppdage. Forfatterne observerer at 'nesten alle 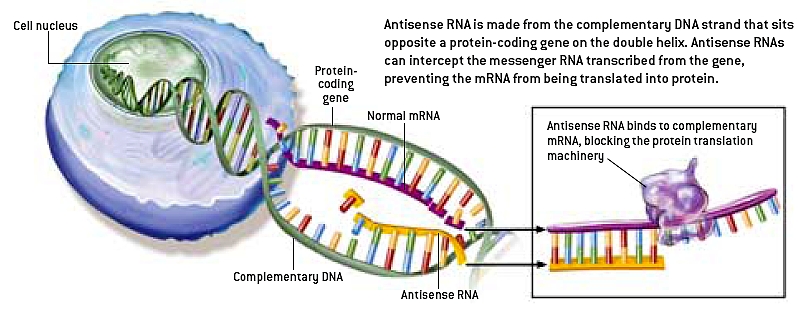 pseudogener som utviser betydelig biologisk aktivitet, uttrykkes i spesifikt vev eller cellelinjer', noe som betyr at bare bestemte vev eller cellelinjer kan bruke et gitt pseudogen til noen funksjon. I tillegg er det vanskelig å oppdage funksjon for pseudogener fordi vi har manglet forskningsverktøyene til å forstå hvordan de påvirker genuttrykk. Artikkelen forutser at 'flere og flere funksjonelle pseudogener vil bli oppdaget når nye biologiske teknologier utvikles i fremtiden', og konkluderer med 'Studien av funksjonelle pseudogener er bare i begynnelsen.' (197) I virkeligheten rapporterte to ledende biologer som skriver i Annual Review of Genetics at "pseudogener som har blitt hensiktsmessig undersøkt, ofte utfører funksjonelle roller." (198)
pseudogener som utviser betydelig biologisk aktivitet, uttrykkes i spesifikt vev eller cellelinjer', noe som betyr at bare bestemte vev eller cellelinjer kan bruke et gitt pseudogen til noen funksjon. I tillegg er det vanskelig å oppdage funksjon for pseudogener fordi vi har manglet forskningsverktøyene til å forstå hvordan de påvirker genuttrykk. Artikkelen forutser at 'flere og flere funksjonelle pseudogener vil bli oppdaget når nye biologiske teknologier utvikles i fremtiden', og konkluderer med 'Studien av funksjonelle pseudogener er bare i begynnelsen.' (197) I virkeligheten rapporterte to ledende biologer som skriver i Annual Review of Genetics at "pseudogener som har blitt hensiktsmessig undersøkt, ofte utfører funksjonelle roller." (198)
Mange evolusjonære biologer er knyttet til oppfatningen at våre genom er fulle av søppel, og motstår fortolkningen at nesten alt DNA har funksjon. Faktisk lærer en lærebok for 2012 at "Over halvparten av genomet er sammensatt av verken gener eller levninger av menneskelige gener eller regulatoriske regioner. I stedet består den av parasitt-lignende segmenter av DNA ... " (199) I mellomtiden fortsetter bevisene i motsatt retning. Mens mye gjenstår å bli lært om virkemåten til vårt genom, er forskningstendensen utvetydig. Jo mer vi studerer ikke-kodende DNA, jo mer finner vi bevis for utstrakt funksjon.
 Referanser:
Referanser:
[157.] Horatio Hackett Newman, quoted in The World’s Most Famous Court Trial: Tennessee Evolution Case, 2nd ed. (Dayton, TN: Bryan College, 1990), 268. See also Robert Wiedersheim, The Structure of Man: An Index to His Past History (London: MacMillan and Co, 1895; reprinted by Kessinger, 2007).
[158.] Laura Spinney, "Vestigial organs: Remnants of evolution," New Scientist, 2656 (May 14, 2008), at http://www.newscientist.com/article/mg19826562.100-vestigial-organs-remnants-of-evolution.html
[159.] Sylvia S. Mader, Inquiry into Life, 10th ed. (McGraw Hill, 2003), 293.
[160.] Laura Spinney, "The Five things humans no longer need," New Scientist (May 19, 2008), at http://www.newscientist.com/article/dn13927-five-things-humans-no-longer-need.html
[161.] Douglas Theobald, "29+ Evidences for Macroevolution," TalkOrigins.org, at http://www.talkorigins.org/faqs/comdesc/section2.html
[162.] See Loren G. Martin, "What is the function of the human appendix? Did it once have a purpose that has since been lost?," Scientific American (October, 21, 1999)
[163.] William Parker quoted in Charles Q. Choi, "The Appendix: Useful and in Fact Promising," LiveScience (August 24, 2009).
[164.] Miller, "Life’s Grand Design," 24-32. Miller cites "orphaned genes" but these are not normally understood to be functionless genes. Rather, orphan genes are functional genes that have no known homology to any other gene. Such orphan genes provide evidence for intelligent design because there is no plausible source for their information.
[165.] Richard Dawkins, "The Information Challenge," The Skeptic, 18 (December, 1998).
[166.] Francis Collins, The Language of God: A Scientist Presents Evidence for Belief (New York: Free Press, 2006), 136-37. [167.] Ibid., pp. 134-137.
[168.] Richard Sternberg, "On the Roles of Repetitive DNA Elements in the Context of a Unified Genomic- Epigenetic System," Annals of the New York Academy of Sciences, 981 (2002): 154-88. [169.] Ibid. [170.] Ibid.
[171.] Tammy A. Morrish, Nicolas Gilbert, Jeremy S. Myers, Bethaney J. Vincent, Thomas D. Stamato, Guillermo E. Taccioli, Mark A. Batzer, and John V. Mora "DNA repair mediated by endonuclease-independent LINE-1 retrotransposition," Nature Genetics, 31 (June, 2002): 159-65.
[172.] Galit Lev-Maor, Rotem Sorek, Noam Shomron, and Gil Ast, "The birth of an alternatively spliced exon: 3′ splice-site selection in Alu exons," Science, 300 (May 23, 2003): 1288-91; Wojciech Makalowski, "Not junk after all," Science, 300 (May 23, 2003): 1246-47.
[173.] Nurit Paz-Yaacova, Erez Y. Levanonc, Eviatar Nevod, Yaron Kinare, Alon Harmelinf, Jasmine Jacob-Hirscha, Ninette Amariglioa, Eli Eisenbergg, and Gideon Rechavi, "Adenosine-to-inosine RNA editing shapes transcriptome diversity in primates," Proceedings of the National Academy of Sciences USA, 107 (July 6, 2010): 12174-79.
[174.] Morrish et al., "DNA repair mediated by endonuclease-independent LINE-1 retrotransposition," 159-65; Annie Tremblay, Maria Jasin, and Pierre Chartrand, "A Double-Strand Break in a Chromosomal LINE Element Can Be Repaired by Gene Conversion with Various Endogenous LINE Elements in Mouse Cells," Molecualr and Cellular Biology, 20 (January, 2000): 54-60; Ulf Grawunder, Matthias Wilm, Xiantuo Wu, Peter Kulesza, Thomas E. Wilson, Matthias Mann, and Michael R. Lieber, "Activity of DNA ligase IV stimulated by complex formation with XRCC4 protein in mammalian cells," Nature, 388 (July 31, 1997): 492-95; Thomas E. Wilson, Ulf Grawunder, and Michael R. Lieber, "Yeast DNA ligase IV mediates non-homologous DNA end joining," Nature, 388 (July 31, 1997): 495-98.
[175.] Richard Sternberg and James A. Shapiro, "How repeated retroelements format genome function," Cytogenetic and Genome Research, 110 (2005): 108-16.
[176.] Jeffrey S. Han, Suzanne T. Szak, and Jef D. Boeke, "Transcriptional disruption by the L1 retrotransposon and implications for mammalian transcriptomes," Nature, 429 (May 20, 2004): 268-74; Bethany A. Janowski, Kenneth E. Huffman, Jacob C. Schwartz, Rosalyn Ram, Daniel Hardy, David S. Shames, John D. Minna, and David R. Corey, "Inhibiting gene expression at transcription start sites in chromosomal DNA with antigene RNAs," Nature Chemical Biology, 1 (September, 2005): 216-22; J. A. Goodrich, and J. F. Kugel, "Non-coding-RNA regulators of RNA polymerase II transcription," Nature Reviews Molecular and Cell Biology, 7 (August, 2006): 612-16; L.C. Li, S. T. Okino, H. Zhao, H., D. Pookot, R. F. Place, S. Urakami, H. Enokida, and R. Dahiya, "Small dsRNAs induce transcriptional activation in human cells," Proceedings of the National Academy of Sciences USA, 103 (November 14, 2006): 17337-42; A. Pagano, M. Castelnuovo, F. Tortelli, R. Ferrari, G. Dieci, and R. Cancedda, "New small nuclear RNA gene-like transcriptional units as sources of regulatory transcripts," PLoS Genetics, 3 (February, 2007): e1; L. N. van de Lagemaat, J. R. Landry, and D. L. Mager, P. Medstrand, "Transposable elements in mammals promote regulatory variation and diversification of genes with specialized functions," Trends in Genetics, 19 (October, 2003): 530-36; S. R. Donnelly, T. E. Hawkins, and S. E. Moss, "A Conserved nuclear element with a role in mammalian gene regulation," Human Molecular Genetics, 8 (1999): 1723-28; C. A. Dunn, P. Medstrand, and D. L. Mager, "An endogenous retroviral long terminal repeat is the dominant promoter for human B1,3- galactosyltransferase 5 in the colon," Proceedings of the National Academy of Sciences USA, 100 (October 28, 2003):12841-46; B. Burgess-Beusse, C. Farrell, M. Gaszner, M. Litt, V. Mutskov, F. Recillas-Targa, M. Simpson, A. West, and G. Felsenfeld, "The insulation of genes from external enhancers and silencing chromatin," Proceedings of the National Academy of Sciences USA, 99 (December 10, 2002): 16433-37; P. Medstrand, Josette-Renée Landry, and D. L. Mager, "Long Terminal Repeats Are Used as Alternative Promoters for the Endothelin B Receptor and Apolipoprotein C-I Genes in Humans," Journal of Biological Chemistry, 276 (January 19, 2001): 1896-1903; L. Mariño-Ramíreza, K.C. Lewisb, D. Landsmana, and I.K. Jordan, "Transposable elements donate lineage-specific regulatory sequences to host genomes," Cytogenetic and Genome Research, 110 (2005):333-41.
[177.] S. Henikoff, K. Ahmad, H. and S. Malik "The Centromere Paradox: Stable Inheritance with Rapidly Evolving DNA," Science, 293 (August 10, 2001): 1098-1102; C. Bell, A. G. West, and G. Felsenfeld, "Insulators and Boundaries: Versatile Regulatory Elements in the Eukaryotic Genome," Science, 291 (January 19, 2001): 447-50; M.-L. Pardue and P.G. DeBaryshe, "Drosophila telomeres: two transposable elements with important roles in chromosomes," Genetica, 107 (1999): 189-96; S. Henikoff, "Heterochromatin function in complex genomes," Biochimica et Biophysica Acta, 1470 (February, 2000): O1-O8; L. M.Figueiredo, L. H. Freitas-Junior, E. Bottius, Jean-Christophe Olivo-Marin, and A. Scherf, "A central role for Plasmodium falciparum subtelomeric regions in spatial positioning and telomere length regulation," The EMBO Journal, 21 (2002): 815-24; Mary G. Schueler, Anne W. Higgins, M. Katharine Rudd, Karen Gustashaw, and Huntington F. Willard, "Genomic and Genetic Definition of a Functional Human Centromere," Science, 294 (October 5, 2001): 109-15.
[178.] Ling-Ling Chen, Joshua N. DeCerbo, and Gordon G. Carmichael, "Alu element-mediated gene silencing," The EMBO Journal 27 (2008): 1694-1705; Jerzy Jurka, "Evolutionary impact of human Alu repetitive elements," Current Opinion in Genetics & Development, 14 (2004): 603-8; G. Lev-Maor et al. "The birth of an alternatively spliced exon: 3′ splice-site selection in Alu exons," 1288-91; E. Kondo-Iida, K. Kobayashi, M. Watanabe, J. Sasaki, T. Kumagai, H. Koide, K. Saito, M. Osawa, Y. Nakamura, and T. Toda, "Novel mutations and genotype-phenotype relationships in 107 families with Fukuyama-type congenital muscular dystrophy (FCMD)," Human Molecular Genetics, 8 (1999): 2303-09; John S. Mattick and Igor V. Makunin, "Non-coding RNA," Human Molecular Genetics, 15 (2006): R17-R29.
[179.] M. Mura, P. Murcia, M. Caporale, T. E. Spencer, K. Nagashima, A. Rein, and M. Palmarini, "Late viral interference induced by transdominant Gag of an endogenous retrovirus," Proceedings of the National Academy of Sciences USA, 101 (July 27, 2004): 11117-22; M. Kandouz, A. Bier, G. D Carystinos, M. A Alaoui-Jamali, and G. Batist, "Connexin43 pseudogene is expressed in tumor cells and inhibits growth," Oncogene, 23 (2004):4763-70.
[180.] K. A. Dunlap, M. Palmarini, M. Varela, R. C. Burghardt, K. Hayashi, J. L. Farmer, and T. E. Spencer, "Endogenous retroviruses regulate periimplantation placental growth and differentiation," Proceedings of the National Academy of Sciences USA, 103 (September 26, 2006):14390-95; L. Hyslop, M. Stojkovic, L. Armstrong, T. Walter, P. Stojkovic, S. Przyborski, M. Herbert, A. Murdoch, T. Strachan, and M. Lakoa, "Downregulation of NANOG Induces Differentiation of Human Embryonic Stem Cells to Extraembryonic Lineages," Stem Cells, 23 (2005): 1035-43; E. Peaston, A. V. Evsikov, J. H. Graber, W. N. de Vries, A. E. Holbrook, D. Solter, and B. B. Knowles, "Retrotransposons Regulate Host Genes in Mouse Oocytes and Preimplantation Embryos," Developmental Cell, 7 (October, 2004): 597-606.
[181.] Richard Sternberg and James A. Shapiro, "How repeated retroelements format genome function," Cytogenetic and Genome Research, 110 (2005): 108-16.
[182.] The ENCODE Project Consortium, "An integrated encyclopedia of DNA elements in the human genome," Nature, 489:57-74 (September 6, 2012).
[183.] Ewan Birney, quoted in Ed Yong, "ENCODE: the rough guide to the human genome," Discover Magazine (September 5, 2012), at http://blogs.discovermagazine.com/notrocketscience/2012/09/05/encode-the-rough-guide-to-the-human-genome/
[184.] Tom Gingeras, quoted in Ed Yong, "ENCODE: the rough guide to the human genome," Discover Magazine (September 5, 2012), at http://blogs.discovermagazine.com/notrocketscience/2012/09/05/encode-the-rough-guide-to-the-human-genome/
[185.] Joseph R. Ecker, "Serving up a genome feast," Nature, 489:52-55 (September 6, 2012).
[186.] Ed Yong, "ENCODE: the rough guide to the human genome," Discover Magazine (September 5, 2012), at http://blogs.discovermagazine.com/notrocketscience/2012/09/05/encode-the-rough-guide-to-the-human-genome/
[187.] Makalowski, "Not Junk After All," 1246-47. [188.] Ibid.
[189.] David R. Kelley and John L. Rinn, "Transposable elements reveal a stem cell specific class of long noncoding RNAs," Genome Biology, 13:R107 (2012).
[190.] Laura Poliseno, "Pseudogenes: Newly Discovered Players in Human Cancer," Science Signaling, 5 (242) (September 18, 2012). [191.] Ibid.
[192.] See for example D. Zheng and M. B. Gerstein, "The ambiguous boundary between genes and pseudogenes: the dead rise up, or do they?," Trends in Genetics, 23 (May, 2007): 219-24; S. Hirotsune et al., "An expressed pseudogene regulates the messenger-RNA stability of its homologous coding gene," Nature, 423 (May 1, 2003): 91-96; O. H. Tam et al., "Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes," Nature, 453 (May 22, 2008): 534-38; D. Pain et al., "Multiple Retropseudogenes from Pluripotent Cell-specific Gene Expression Indicates a Potential Signature for Novel Gene Identification," The Journal of Biological Chemistry, 280 (February 25, 2005):6265-68; J. Zhang et al., "NANOGP8 is a retrogene expressed in cancers," FEBS Journal, 273 (2006): 1723-30.
[193.] The ENCODE Project Consortium, "An integrated encyclopedia of DNA elements in the human genome," Nature, 489:57-74 (September 6, 2012).
[194.] Ryan Charles Pink, Kate Wicks, Daniel Paul Caley, Emma Kathleen Punch, Laura Jacobs, and David Paul Francisco Carter, "Pseudogenes: Pseudo-functional or key regulators in health and disease?," RNA, 17 (2011): 792-98.
[195.] Yan-Zi Wen, Ling-Ling Zheng, Liang-Hu Qu, Francisco J. Ayala and Zhao-Rong Lun, "Pseudogenes are not pseudo any more," RNA Biology, 9(1):27-32 (January, 2012). [196.] Ibid. [197.] Ibid.
[198.] Evgeniy S. Balakirev and Francisco J. Ayala, "Pseudogenes, Are They ‘Junk’ or Functional DNA?," Annual Review of Genetics, 37 (2003): 123-51.
[199.] Carl Zimmer and Douglas Emlen, Evolution: Making Sense of Life, p. 132 (Roberts and Company, 2012).
Oversettelse og bilder ved Asbjørn E. Lund
 I flere tiår har evolusjonister hevdet at kroppene og genomene våre er fulle av ubrukelige deler og genetisk materiale - 'vestigiale' organer - som viser at livet er et resultat av eoner av ikke-styrt evolusjon. Under Scopes-rettssaken i 1925 hevdet evolusjonærbiologen Horatio Hackett Newman at det er over 180 vestigiale organer og strukturer i menneskekroppen, 'tilstrekkelig til å gjøre en mann til et veritabelt vandrende museum av antikviteter.' (157)
I flere tiår har evolusjonister hevdet at kroppene og genomene våre er fulle av ubrukelige deler og genetisk materiale - 'vestigiale' organer - som viser at livet er et resultat av eoner av ikke-styrt evolusjon. Under Scopes-rettssaken i 1925 hevdet evolusjonærbiologen Horatio Hackett Newman at det er over 180 vestigiale organer og strukturer i menneskekroppen, 'tilstrekkelig til å gjøre en mann til et veritabelt vandrende museum av antikviteter.' (157) Halebenet (coccyx ): Mange evolusjonister hevder fortsatt at dette 'henger igjen' fra haler til våre antatte primater forfedre, (160), men det er faktisk en viktig del av skjelettet vårt, som brukes til å feste muskler, sener og leddbånd som støtter beinene i bekkenet vårt.
Halebenet (coccyx ): Mange evolusjonister hevder fortsatt at dette 'henger igjen' fra haler til våre antatte primater forfedre, (160), men det er faktisk en viktig del av skjelettet vårt, som brukes til å feste muskler, sener og leddbånd som støtter beinene i bekkenet vårt. Mange forskere som tjener som talsmenn for evolusjonær biologi har hevdet at dette beviset 'lukker saken', som bevis for darwinistisk evolusjon:
Mange forskere som tjener som talsmenn for evolusjonær biologi har hevdet at dette beviset 'lukker saken', som bevis for darwinistisk evolusjon: Sternberg, sammen med University of Chicago genetikeren James Shapiro, forutsa i 2005 i tidsskriftet 'Cytogenetic and Genome Research' at 'en dag vil vi tenke på det som pleide å bli kalt 'junk DNA' som en kritisk del av virkelig 'ekspert' cellulære kontroll-regimer'. (181)
Sternberg, sammen med University of Chicago genetikeren James Shapiro, forutsa i 2005 i tidsskriftet 'Cytogenetic and Genome Research' at 'en dag vil vi tenke på det som pleide å bli kalt 'junk DNA' som en kritisk del av virkelig 'ekspert' cellulære kontroll-regimer'. (181) Ewan Birney, ENCODEs ledende analysekoordinator kommenterte i Discover Magazine at siden ENCODE så på bare 147 typer celler, og menneskekroppen har noen få tusen, "Det er sannsynlig at 80 prosent vil øke til 100 prosent." (183) I samme artikkel sitert Tom Gingeras, en seniorforsker med ENCODE, og bemerker at "nesten hvert nukleotide er knyttet til en eller annen funksjon, og vi vet nå hvor de er, hva som binder til dem, hva deres relasjoner er, og mer." En annen Nature kommentar bemerket at "80% av genomet inneholder elementer knyttet til biokjemiske funksjoner, og senker den allment holdte oppfatningen om at det menneskelige genomet hovedsakelig er 'junk DNA'. (185) Discover Magazine sa det slik: "Hovedpoenget er: Det er ikke" søppel ". (186)
Ewan Birney, ENCODEs ledende analysekoordinator kommenterte i Discover Magazine at siden ENCODE så på bare 147 typer celler, og menneskekroppen har noen få tusen, "Det er sannsynlig at 80 prosent vil øke til 100 prosent." (183) I samme artikkel sitert Tom Gingeras, en seniorforsker med ENCODE, og bemerker at "nesten hvert nukleotide er knyttet til en eller annen funksjon, og vi vet nå hvor de er, hva som binder til dem, hva deres relasjoner er, og mer." En annen Nature kommentar bemerket at "80% av genomet inneholder elementer knyttet til biokjemiske funksjoner, og senker den allment holdte oppfatningen om at det menneskelige genomet hovedsakelig er 'junk DNA'. (185) Discover Magazine sa det slik: "Hovedpoenget er: Det er ikke" søppel ". (186) Faktisk har funksjoner for mange pseudogener blitt oppdaget; (192) ENCODE-prosjektet alene fant over 850 pseudogener som er 'transkribert og forbundet med aktivt kromatin.' (193) Men hva gjør disse pseudogenene? En 2011-artikkel i journalen RNA argumenterer igjen for at de kan regulere uttrykket av gener:
Faktisk har funksjoner for mange pseudogener blitt oppdaget; (192) ENCODE-prosjektet alene fant over 850 pseudogener som er 'transkribert og forbundet med aktivt kromatin.' (193) Men hva gjør disse pseudogenene? En 2011-artikkel i journalen RNA argumenterer igjen for at de kan regulere uttrykket av gener: pseudogener som utviser betydelig biologisk aktivitet, uttrykkes i spesifikt vev eller cellelinjer', noe som betyr at bare bestemte vev eller cellelinjer kan bruke et gitt pseudogen til noen funksjon. I tillegg er det vanskelig å oppdage funksjon for pseudogener fordi vi har manglet forskningsverktøyene til å forstå hvordan de påvirker genuttrykk. Artikkelen forutser at 'flere og flere funksjonelle pseudogener vil bli oppdaget når nye biologiske teknologier utvikles i fremtiden', og konkluderer med 'Studien av funksjonelle pseudogener er bare i begynnelsen.' (197) I virkeligheten rapporterte to ledende biologer som skriver i Annual Review of Genetics at "pseudogener som har blitt hensiktsmessig undersøkt, ofte utfører funksjonelle roller." (198)
pseudogener som utviser betydelig biologisk aktivitet, uttrykkes i spesifikt vev eller cellelinjer', noe som betyr at bare bestemte vev eller cellelinjer kan bruke et gitt pseudogen til noen funksjon. I tillegg er det vanskelig å oppdage funksjon for pseudogener fordi vi har manglet forskningsverktøyene til å forstå hvordan de påvirker genuttrykk. Artikkelen forutser at 'flere og flere funksjonelle pseudogener vil bli oppdaget når nye biologiske teknologier utvikles i fremtiden', og konkluderer med 'Studien av funksjonelle pseudogener er bare i begynnelsen.' (197) I virkeligheten rapporterte to ledende biologer som skriver i Annual Review of Genetics at "pseudogener som har blitt hensiktsmessig undersøkt, ofte utfører funksjonelle roller." (198) Referanser:
Referanser: